聚类分析之处理无类标数据
发表于|更新于
|字数总计:3.7k|阅读时长:15分钟|阅读量:
前面几章中,我们使用的数据都是事先已经直到预测结果的,即训练数据中已提供了数据的类标。在本章中,我们转而研究聚类分析,它是一种无监督学习技术,可以在事先不知道正确结果的情况下,发现数据本身所蕴含的结构等信息。
使用k-means算法对相似对象进行分组
本节讨论最流行的聚类算法:k-means算法,它在学术邻域及业界都得到了广泛应用。聚类是一种可以找到相似对象群组的技术,与组间对象相比,组内对象之间具有更高的相似度。
尽管k-means算法适用于高维数据,但出于可视化需要,我们使用一个二维数据集的例子演示:
1
2
3
4
5
6
7
8
9
10
11
12
| from sklearn.datasets import make_blobs
import matplotlib.pyplot as plt
X, y = make_blobs(n_samples=150,
n_features=2,
centers=3,
cluster_std=0.5,
shuffle=True,
random_state=0)
plt.scatter(X[:, 0], X[:, 1], c='blue', marker='o', s=50)
plt.grid()
plt.show()
|
图像如下:

k-means算法具体有四个步骤:
- 从样本点随机选择k个点作为初始簇中心
- 将每个样本点划分到距离它最近的中心点$ \mu^j, j\in{1,\cdots ,k} $所代表的簇中
- 用各簇中所有样本的中心点替代原有的中心点
- 重复步骤2和3,直到中心点不变或者达到预定迭代次数时,算法终止
度量对象之间的相似性可以用欧几里得距离的平方:
$$
d(x, y)^2 = \sum_{j=1}^{m}(x_j-y_j)^2=||x-y||^2_2
$$
基于欧几里得标准,我们可以将k-means算法描述为一个简单的优化问题,也就是使得簇内误差平方和(SSE)最小:
$$
SSE = \sum_{j=1}^n\sum_{j=1}^{k}w^{i,j}=||x^i-\mu^j||_2^2
$$
现在借助scikit-learn中的KMeans类将k-means算法应用于我们的示例数据集:
1
2
3
4
5
6
7
8
| from sklearn.cluster import KMeans
km = KMeans(n_clusters=3,
init='random',
n_init=10,
max_iter=300,
tol=1e-04,
random_state=0)
y_km = km.fit_predict(X)
|
在k-means算法的某次迭代中,可能会发生无法收敛的问题,特别是我们设置了较大的max_iter。解决这个问题的方法是为tol参数设置一个较大的值,上述容忍度为1e-04。
k-means++
我们讨论了经典的k-means算法,它使用随机点作为初始中心点,但是初始中心点选择不当,就会导致收敛速度慢的问题。解决此问题的方法是在数据集上多次运行k-mean算法,并且根据SSE选择性能更好的模型。另外一种方案是使用k-means++算法让初始中心点彼此尽可能远离,相比传统的k-means算法,它能够产生更好的结果。k-means++算法的初始化过程如下:
- 初始化一个空的集合M,用于存储选定的k个中心点
- 从输入样本中随机选定第一个中心点$ \mu^j $,并且将其加入到集合M中
- 对于集合M之外的任一样本点$ x^i $,通过计算找到与其平方距离最小的样本$ d(x^i, M)^2 $
- 使用加权概率分布$ \frac{d(\mu, M)^2}{\sum_id(x^i, M)^2} $来随机选择下一个中心点$ \mu^p $
- 重复步骤2,3,直到选定k个中心点
- 基于选定的中心点执行k-means算法
现在对k-means算法的结果做可视化展示:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
| plt.scatter(X[y_km==0, 0],
X[y_km==0, 1],
s=50,
c='lightgreen',
marker='s',
label='cluster 1')
plt.scatter(X[y_km==1, 0],
X[y_km==1, 1],
s=50,
c='orange',
marker='o',
label='cluster 2')
plt.scatter(X[y_km==2, 0],
X[y_km==2, 1],
s=50,
c='lightblue',
marker='v',
label='cluster 3')
plt.scatter(km.cluster_centers_[:, 0],
km.cluster_centers_[:, 1],
s=250,
marker='*',
c='red',
label='centroids')
plt.legend()
plt.grid()
plt.show()
|
图像如下:

散点图显示的结果中3个中心点位于各个簇的中心,分组结果看起来是合理的。
k-means算法的一个缺点是我们必须先指定一个簇数量k,但是在实际应用中,簇数量并不总是显而易见的。
硬聚类与软聚类
硬聚类指每个样本只能划至一个簇的算法,如k-means算法;软聚类算法可以将样本划分到一个或多个簇,如FCM算法。
FCM算法和k-means算法十分相似,k-means算法某个样本预测结果是$ [0,1,0] $,表明该样本属于簇2。FCM中可以允许预测的结果中含有分数,如$ [0.7, 0.2, 0.1] $表明该样本属于簇1的概率是0.7,簇2的概率是0.2。FCM的步骤如下:
- 指定k个中心点,并随机将样本点划分至某个簇
- 计算各个簇的中心$ \mu^j ,j\in{1, \cdots,k}$
- 更新各样本点所属簇的成员隶属度
- 重复步骤2,3,直到各个样本点所属簇成员隶属度不变或者是达到最大的迭代次数
FCM的目标函数如下:
$$
J_m = \sum_{i=1}^{n}\sum_{j=1}^{m}w^m(i,j)||x^i-\mu^j||^2_2
$$
使用肘方法确定簇的最佳数量
簇内误差平方和可以通过inertia访问,基于簇内误差平方和,我们可以使用图形工具,即所谓的肘方法,针对特定任务估计出最优的簇数量k。
1
2
3
4
5
6
7
8
9
10
11
12
13
| distortions = []
for i in range(1, 11):
km = KMeans(n_clusters=i,
init='k-means++',
n_init=10,
max_iter=300,
random_state=0)
km.fit(X)
distortions.append(km.inertia_)
plt.plot(range(1, 11), distortions, marker='o')
plt.xlabel('Number of clusters')
plt.ylabel('Distortion')
plt.show()
|
图像如下:

如图,当k=3时团呈现肘形,这表明对于此数据来说,k=3是一个好的选择。
通过轮廓图定量分析聚类质量
另外一种聚类质量的评估方法时轮廓分析,此方法用于k-means算法之外的其他聚类方法。我们通过以下步骤计算轮廓系数:
将某样本$ x^i $与簇内其他点之间的平均距离看作是簇的内聚度$ a^i $
将样本$ x^i $与其最近簇中所有点之间的平均距离看作是与下一最近簇的分离度$ b^i $
轮廓系数如下:
$$
s^i = \frac{b^i - a^i}{max{b^i, a^i}}
$$
可以发现,理想的轮廓系数时1,轮廓系数代码如下:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
| km = KMeans(n_clusters=3,
init='k-means++',
n_init=10,
max_iter=300,
tol=1e-04,
random_state=0)
y_km = km.fit_predict(X)
import numpy as np
from matplotlib import cm
from sklearn.metrics import silhouette_samples
cluster_labels = np.unique(y_km)
n_clusters = cluster_labels.shape[0]
silhouette_vals = silhouette_samples(X,
y_km,
metric='euclidean')
y_ax_lower, y_ax_upper = 0, 0
yticks = []
for i, c in enumerate(cluster_labels):
c_silhouette_vals = silhouette_vals[y_km == c]
c_silhouette_vals.sort()
y_ax_upper += len(c_silhouette_vals)
color = cm.jet(float(i) / n_clusters)
plt.barh(range(y_ax_lower, y_ax_upper),
c_silhouette_vals,
height=1.0,
edgecolor='none',
color=color)
yticks.append((y_ax_lower + y_ax_upper) / 2.)
y_ax_lower += len(c_silhouette_vals)
silhouette_avg = np.mean(silhouette_vals)
plt.axvline(silhouette_avg,
color="red",
linestyle="--")
plt.yticks(yticks, cluster_labels + 1)
plt.ylabel('Cluster')
plt.xlabel('Silhouette coefficient')
plt.show()
|
图像如下:

从上图可见,轮廓系数未接近0点,此指标显示聚类效果不错。为了解聚类效果不佳的轮廓图的形状,我们使用两个中心点来初始化k-means算法:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
| km = KMeans(n_clusters=2,
init='k-means++',
n_init=10,
max_iter=300,
tol=1e-04,
random_state=0)
y_km = km.fit_predict(X)
plt.scatter(X[y_km==0, 0],
X[y_km==0, 1],
s=50,
c='lightgreen',
marker='s',
label='cluster 1')
plt.scatter(X[y_km==1, 0],
X[y_km==1, 1],
s=50,
c='orange',
marker='o',
label='cluster 2')
plt.scatter(km.cluster_centers_[:, 0],
km.cluster_centers_[:, 1],
s=250,
marker='*',
c='red',
label='centroids')
plt.legend()
plt.grid()
plt.show()
|
图像如下:

接下来,我们绘制轮廓图来对聚类结果进行评估:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
| cluster_labels = np.unique(y_km)
n_clusters = cluster_labels.shape[0]
silhouette_vals = silhouette_samples(X,
y_km,
metric='euclidean')
y_ax_lower, y_ax_upper = 0, 0
yticks = []
for i, c in enumerate(cluster_labels):
c_silhouette_vals = silhouette_vals[y_km == c]
c_silhouette_vals.sort()
y_ax_upper += len(c_silhouette_vals)
color = cm.jet(float(i) / n_clusters)
plt.barh(range(y_ax_lower, y_ax_upper),
c_silhouette_vals,
height=1.0,
edgecolor='none',
color=color)
yticks.append((y_ax_lower + y_ax_upper) / 2.)
y_ax_lower += len(c_silhouette_vals)
silhouette_avg = np.mean(silhouette_vals)
plt.axvline(silhouette_avg,
color="red",
linestyle="--")
plt.yticks(yticks, cluster_labels + 1)
plt.ylabel('Cluster')
plt.xlabel('Silhouette coefficient')
plt.show()
|
由结果可见,轮廓图由明显不同的长度和宽度,这说明该聚类并非最优结果:

层次聚类
本节中,我们将学习另外一种基于原型的聚类:层次聚类。层次聚类算法的优势在于:他能够使我们绘制出树状图,这有助于我们使用有意义的分类解释聚类结果。层次聚类的另外一个优势在于我们无需指定簇数量。
层次聚类有两种主要方法:凝聚层次聚类和分裂层次聚类。在凝聚层次聚类中,判定簇间距离的两个标准方法分别是单连接和全连接。单连接方法计算每一对簇中最相似两个样本的距离,并且合并距离最近的两个样本所属簇。与之相反,全连接方法是通过比较找到分布于两个簇中最不相似的样本(距离最远的样本),进而完成簇的合并。
本节中,我们主要关注基于全连接方法的凝聚层次聚类,迭代过程如下:
- 计算得到所有样本间的距离矩阵
- 将每个数据点看作是一个单独的簇
- 基于最不相似(距离最远)样本的距离,合并两个最接近的簇
- 更新相似矩阵
- 重复步骤2到4,直到所有样本都合并到一个簇为止
计算距离矩阵的方式如下:
1
2
3
4
5
6
7
8
| import pandas as pd
import numpy as np
np.random.seed(123)
variables = ['X', 'Y', 'Z']
labels = ['ID_0', 'ID_1', 'ID_2', 'ID_3', 'ID_4']
X = np.random.random_sample([5, 3])*10
df = pd.DataFrame(X, columns=variables, index=labels)
df
|
得到的数据如下:
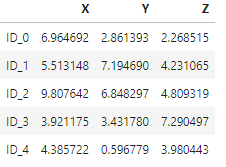
基于距离矩阵进行层次聚类
我们基于SciPy来计算距离矩阵:
1
2
3
| from scipy.spatial.distance import pdist, squareform
row_dist = pd.DataFrame(squareform(pdist(df, metric='euclidean')), columns=labels, index=labels)
row_dist
|
得到的数据如下:
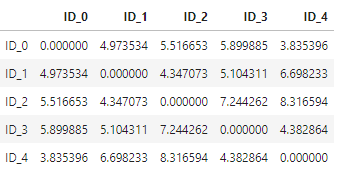
接下来我们使用linkage函数,此函数以全连接作为距离判定标准:
1
2
3
4
| from scipy.cluster.hierarchy import linkage
row_clusters = linkage(df.values, method='complete', metric='euclidean')
pd.DataFrame(row_clusters, columns=['row label 1', 'row label 2', 'distance', 'no. of items'],
index=['cluster %d' % (i+1) for i in range(row_clusters.shape[0])])
|
得到的数据如下:
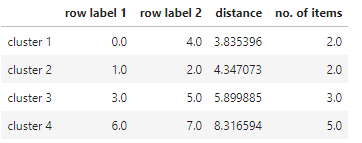
接下来采用树状图的形式对聚类结果进行可视化展示:
1
2
3
4
5
| from scipy.cluster.hierarchy import dendrogram
row_dendr = dendrogram(row_clusters, labels=labels)
plt.tight_layout()
plt.ylabel('Euclidean distance')
plt.show()
|
得到的图像如下:

此树状图采用了凝聚层次聚类合并生成不同簇的过程,从图中可见,首先ID_0和ID_4合并,解下来是ID_1和ID_2合并。
树状图与热度图的关联
实际应用中,层次聚类的树状图与热度图结合使用,本节中我们讨论如何将树状图附加到热度图上:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
| fig = plt.figure(figsize=(8, 8))
axd = fig.add_axes([0.09, 0.1, 0.2, 0.6])
row_dendr = dendrogram(row_clusters, orientation='left')
df_rowclust = df.ix[row_dendr['leaves'][::-1]]
axm = fig.add_axes([0.23, 0.1, 0.6, 0.6])
cax = axm.matshow(df_rowclust, interpolation='nearest', cmap='hot_r')
axd.set_xticks([])
axd.set_yticks([])
for i in axd.spines.values():
i.set_visible(False)
fig.colorbar(cax)
axm.set_xticklabels([''] + list(df_rowclust.columns))
axm.set_yticklabels([''] + list(df_rowclust.index))
plt.show()
|
得到图像可得:

通过scikit-learn进行凝聚聚类
本节使用scikit-learn进行基于凝聚的层次聚类:
1
2
3
4
5
| from sklearn.cluster import AgglomerativeClustering
ac = AgglomerativeClustering(n_clusters=2, affinity='euclidean', linkage='complete')
labels = ac.fit_predict(X)
print('Cluster labels: %s' % labels)
>> Cluster labels: [0 1 1 0 0]
|
通过对簇类标的预测结果进行分析,我们可以看出第一第四第五样本被划分至第一个簇,第二第三样本被划分到第二个簇。
使用DBSCAN划分高密度区域
接下来我们介绍另外一种聚类方法:基于密度空间的聚类算法。在DBSCAN中,基于一下标准,每个样本都被赋予了一个特殊的标签:
- 如果一个点周边的指定半径$ \epsilon $内,其他样本点的数量不小于指定数量(MinPts),则此样本点称为核心点(core point)
- 在指定半径$ \epsilon $内,如果一个点的邻居数量小于MinPts时,但是却包含一个核心点,则此点称为边界点(border point)
- 除了核心点和边界点外的其他样本点称为噪声点(noise point)
完成对核心点,边界点和噪声点的标记后,DBSCAN算法可以总结为两个简单的步骤:
- 基于每个核心点或者一组相连的核心点形成一个单独的簇
- 将每个边界点划分到对应核心点所在的簇中
为了给出一个更能说明问题的例子,我们创建一个半月形的数据集,以及k-means聚类,层次聚类和DBSCAN聚类进行比较,首先得到半月形数据集:
1
2
3
4
| from sklearn.datasets import make_moons
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
plt.scatter(X[:, 0], X[:, 1])
plt.show()
|
得到的图像如下:

下面首先使用前面讨论过的k-means算法和基于全连接的层次聚类算法,算法如下:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
| f, (ax1, ax2) = plt.subplots(1, 2, figsize=(8, 3))
km = KMeans(n_clusters=2, random_state=0)
y_km = km.fit_predict(X)
ax1.scatter(X[y_km==0,0], X[y_km==0,1],
c='lightblue',
edgecolor='black',
marker='o',
s=40,
label='cluster 1')
ax1.scatter(X[y_km==1,0],
X[y_km==1,1],
c='red',
edgecolor='black',
marker='s',
s=40,
label='cluster 2')
ax1.set_title('K-means clustering')
ac = AgglomerativeClustering(n_clusters=2,
affinity='euclidean',
linkage='complete')
y_ac = ac.fit_predict(X)
ax2.scatter(X[y_ac==0,0],
X[y_ac==0,1],
c='lightblue',
edgecolor='black',
marker='o',
s=40,
label='cluster 1')
ax2.scatter(X[y_ac==1,0],
X[y_ac==1,1],
c='red',
edgecolor='black',
marker='s',
s=40,
label='cluster 2')
ax2.set_title('Agglomerative clustering')
plt.legend()
plt.show()
|
得到的图像如下:

可以发现,上述两个算法无法有效分开两个数组。最后我们试一下DBSCAN算法在此数据集上的效果:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
| from sklearn.cluster import DBSCAN
db = DBSCAN(eps=0.2,
min_samples=5,
metric='euclidean')
y_db = db.fit_predict(X)
plt.scatter(X[y_db==0,0],
X[y_db==0,1],
c='lightblue',
edgecolor='black',
marker='o',
s=40,
label='cluster 1')
plt.scatter(X[y_db==1,0],
X[y_db==1,1],
c='red',
edgecolor='black',
marker='s',
s=40,
label='cluster 2')
plt.legend()
plt.show()
|
得到的图像如下:

可以发现,DBSCAN算法可以成功地对半月形数据进行划分,这也是DBSCAN算法的优势。













